HiC热图调整
# 输入文件
xxx.hic:含有全基因组bin之间的互作强度信息,一般互作强度更强烈的contig被聚类到一起
xxx.assembly:连接的染色体信息
上述两个文件由hic辅助组装软件生成(如3Ddna)
# 软件操作
# 载入数据
首先点击File->Open,读入xxx.hic文件,这个时候juicebox就可以展示全基因组的互作热图了
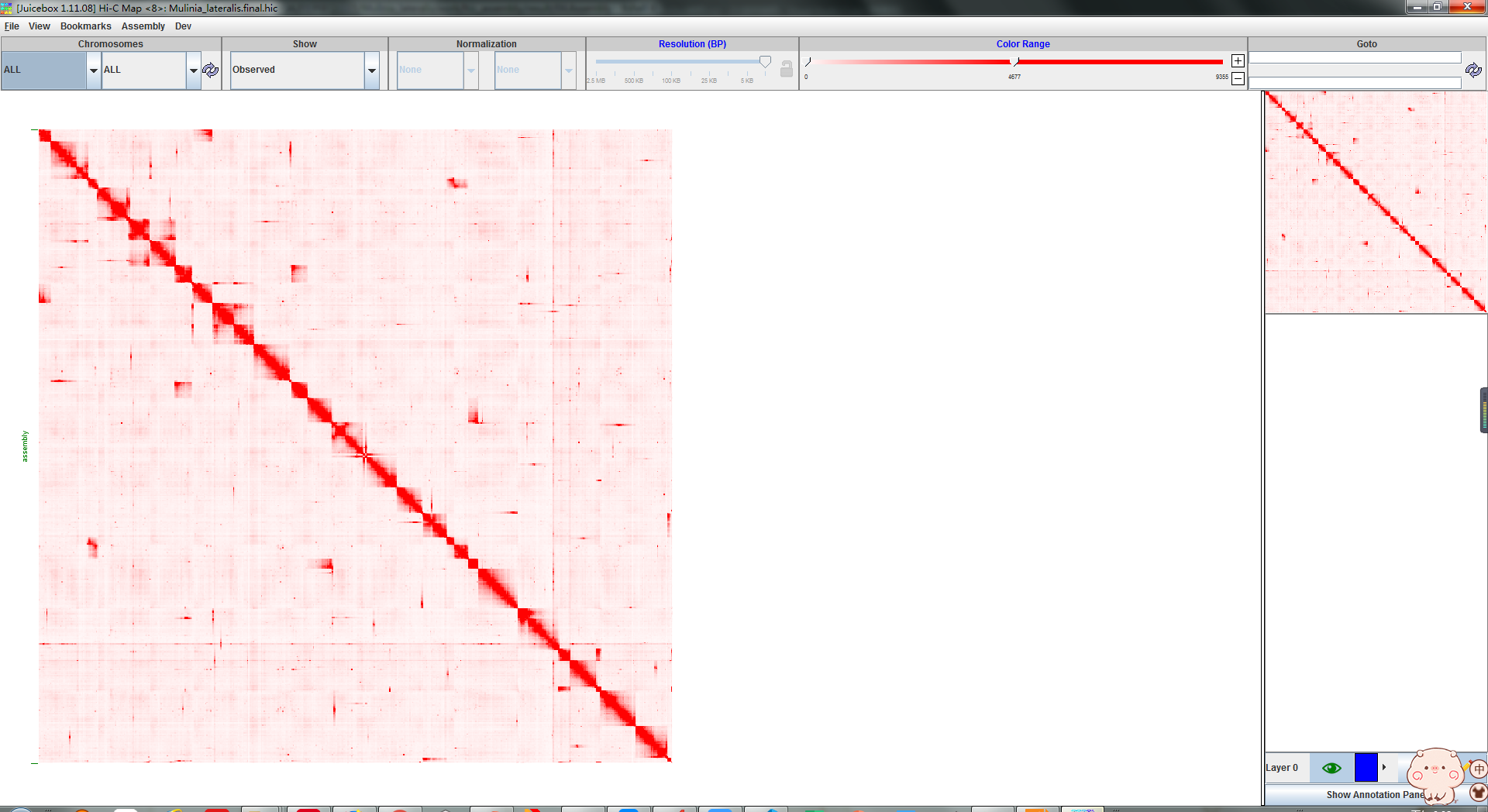 接着点击Assembly->import map assembly,读入xxx.assembly文件,此时juicebox可以展示染色体区域的蓝色边框
接着点击Assembly->import map assembly,读入xxx.assembly文件,此时juicebox可以展示染色体区域的蓝色边框
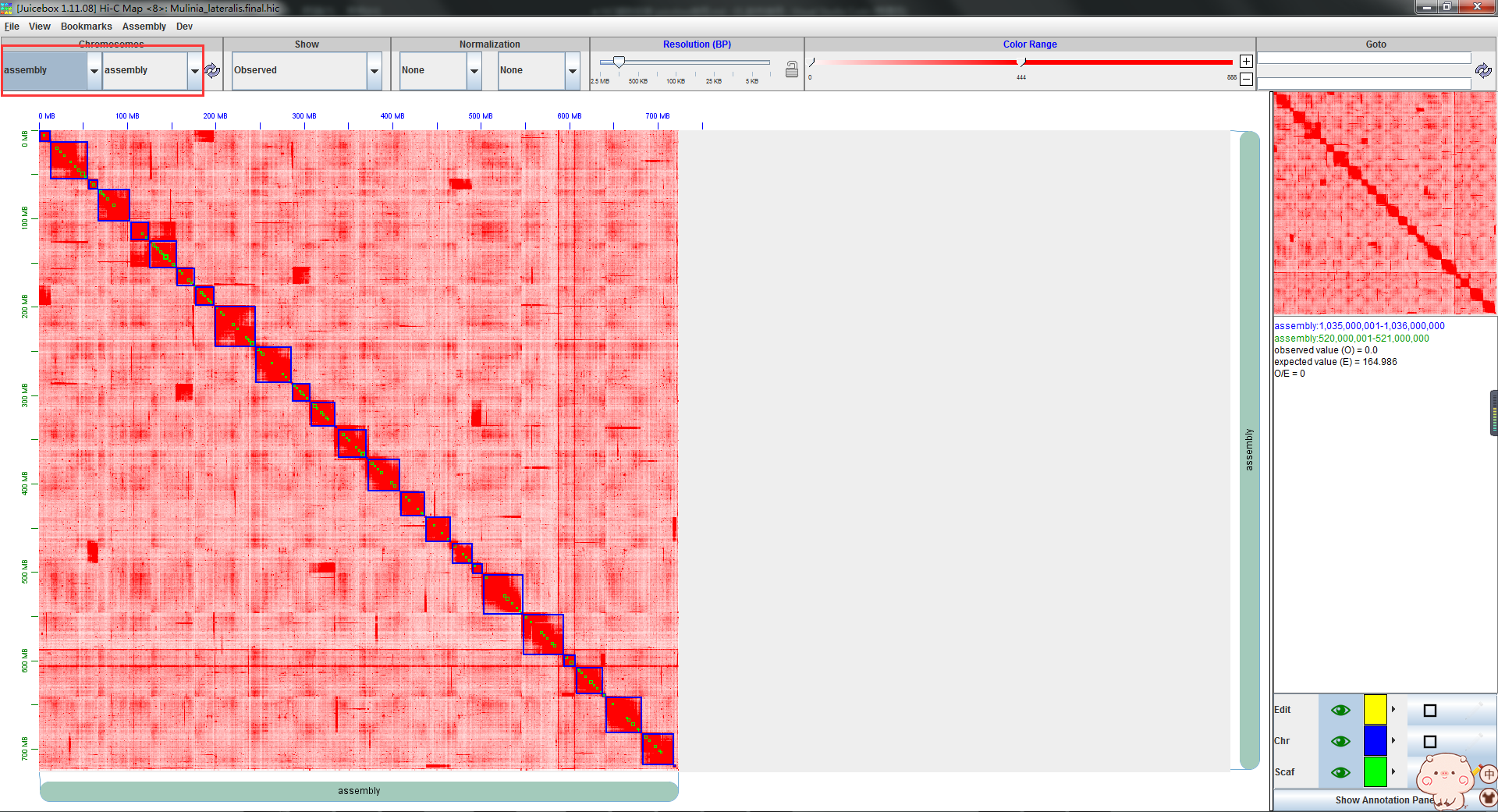
# 染色体结构调整
HiC热图调整的理论基础是:同一条染色体内不同区域的物理位置相较染色体间其它的的区域更接近,染色体内序列位置更近的区域物理位置相对也更近,根据HiC的测序原理可得相邻区域的互作强度更强,反映到热图里则表现为序列位置更近的区域颜色越红。
在进行调整前必须了解研究物种的染色体数,保证调整结果的正确性,本次示例物种的染色体数目为2n=38,由于组装结果是单倍体,即条数应为19条,而上图可见3Ddna结果将全基因组大约分为了24条染色体,下面将进行一步步的切分、连接操作。
# 常用操作方法说明
- 双击放大区域,即调整REsolution(BP)范围;
- 可在菜单栏的normalization中左侧None改为Coverage或者Balanced(类似于均一化的操作),热图一般相对更清晰;
- 右键undo:回溯操作
- shift+A+鼠标左键:选中区域
# 1.旋转
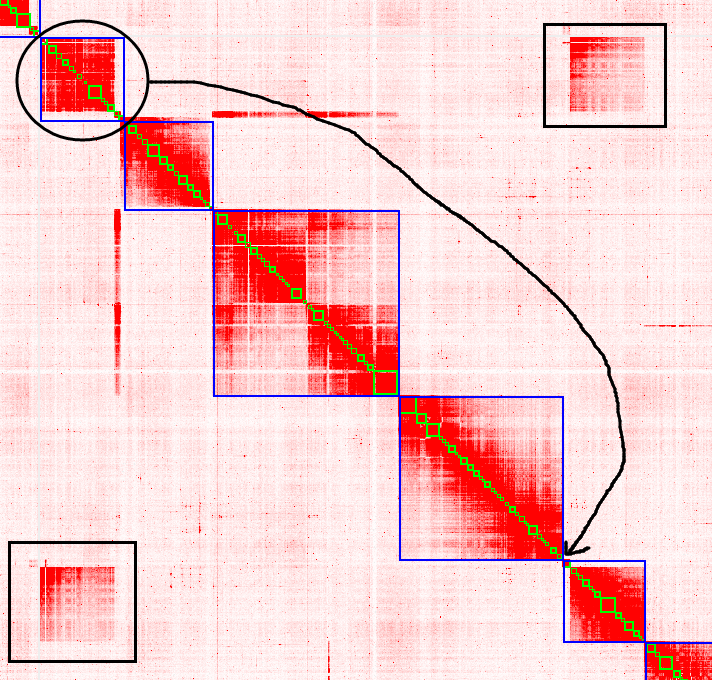
下图中明显可以看到右侧scaffold的右下角区域与左侧scaffold的互作更强,因此需要将其旋转方向,shift+A+鼠标左键选中,将鼠标移动到该scaffold左下角,可以看到鼠标变为旋转标志,左键旋转即可,调整前热图如下:
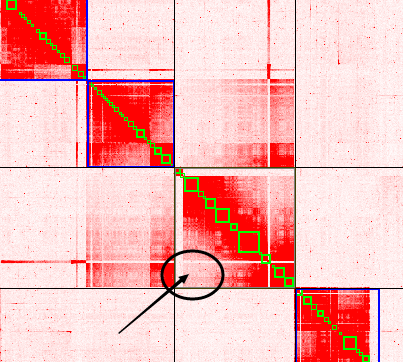
旋转后如下如所示:
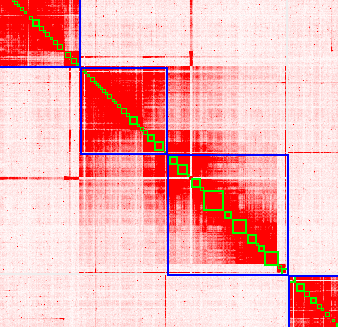
# 2.移动
部分contig/scaffold的位置明显不正确,与热图中相隔较远的区块互作更强,即与该区域应当更近,所以需要移动这部分区域,shift+A+鼠标左键选中需要移动的区域,点击准备插入的蓝框交界处即可(可能需要双击放大插入位置),调整前热图如下所示:
移动后如下图所示:
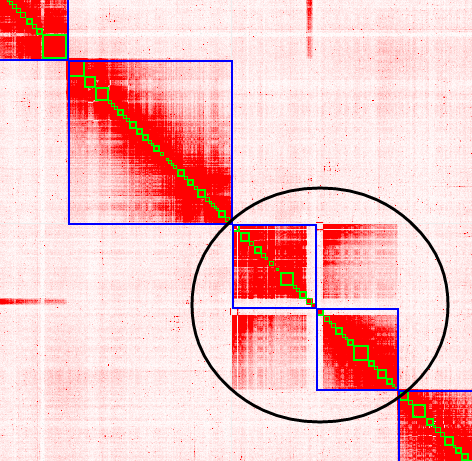
可以看到还需要做旋转操作:
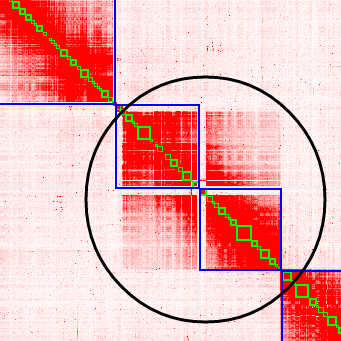
# 3.截断
可以看到上面热图中依然存在异常,部分contig不属于软件标识的scaffold,此时就需要将scaffold截断,将contig放到它应该在的位置,截断操作:双击需调整位置放大热图,将鼠标移动到需截断位置,直至鼠标变为“L”型,左键点击即可:
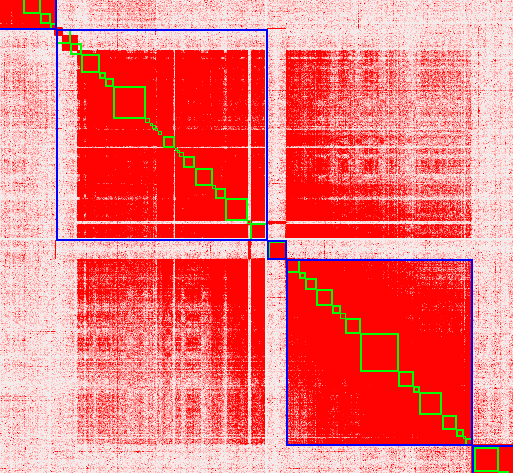
把截断的contig挪走:
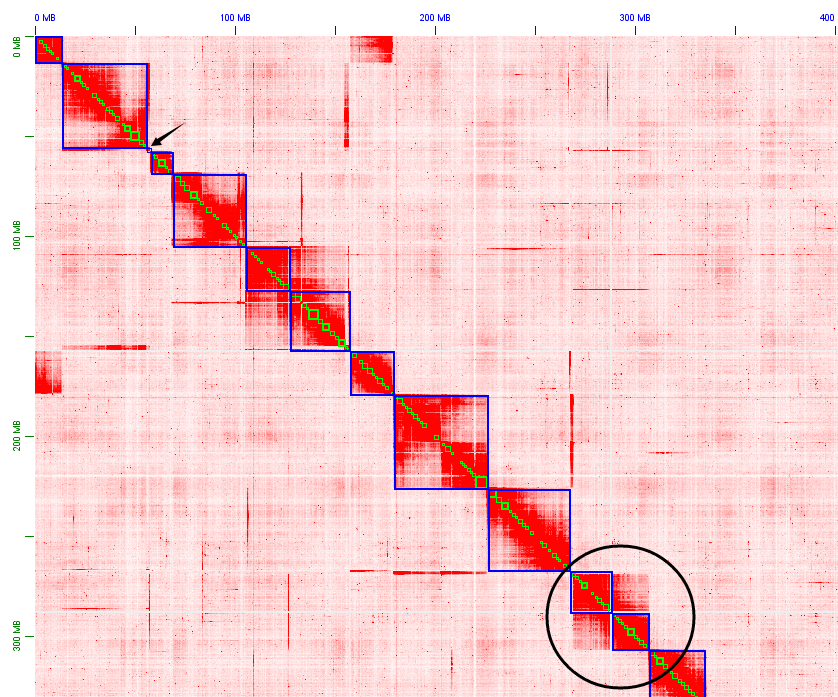
有一些片段非常短,肉眼比较难确定到底和哪个区域互作强,这个时候可以在热图上右键勾上“Enable straight edge”,打开垂直竖直线辅助观察:
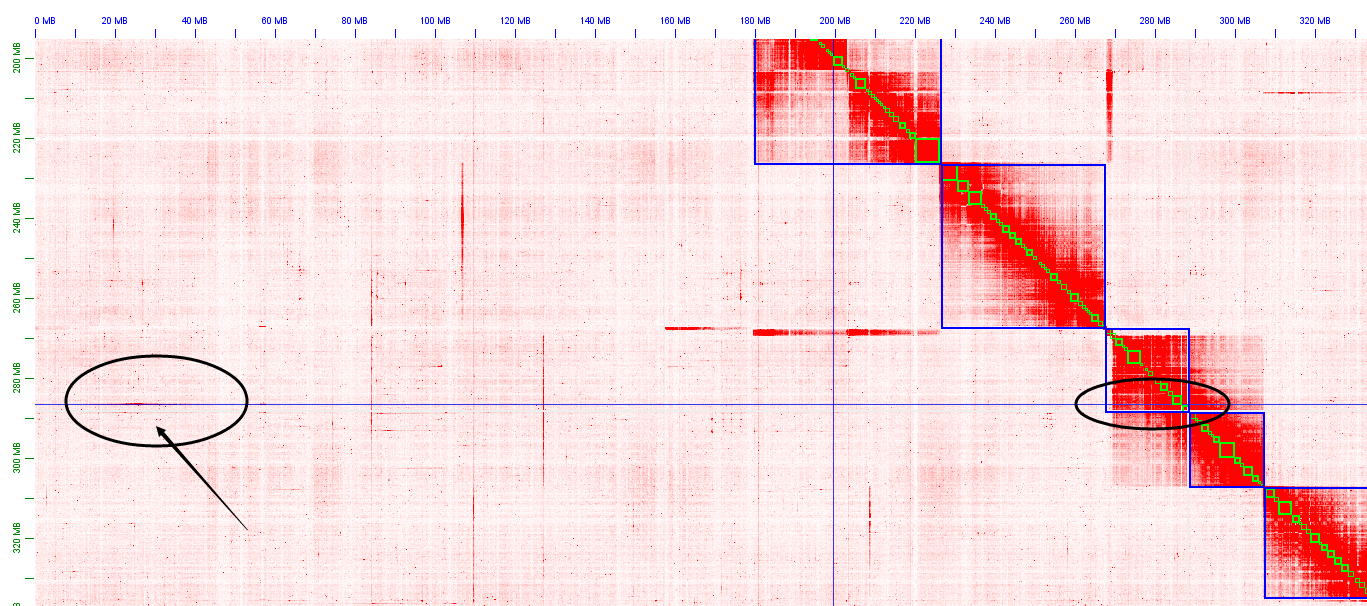 截断contig时需要先shift+A+鼠标左键选中目标contig,将鼠标移动到需截断位置,鼠标会变为剪刀状,左键点击即可,打断前热图如下:
截断contig时需要先shift+A+鼠标左键选中目标contig,将鼠标移动到需截断位置,鼠标会变为剪刀状,左键点击即可,打断前热图如下:
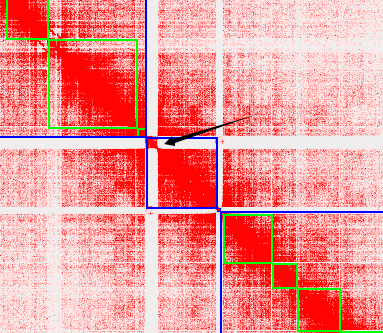
不知道或者无处可放的contig可以选中后,右键菜单点击“Move to debris”将其挪到热图尾部未挂载区域,打断并移走后热图如下:
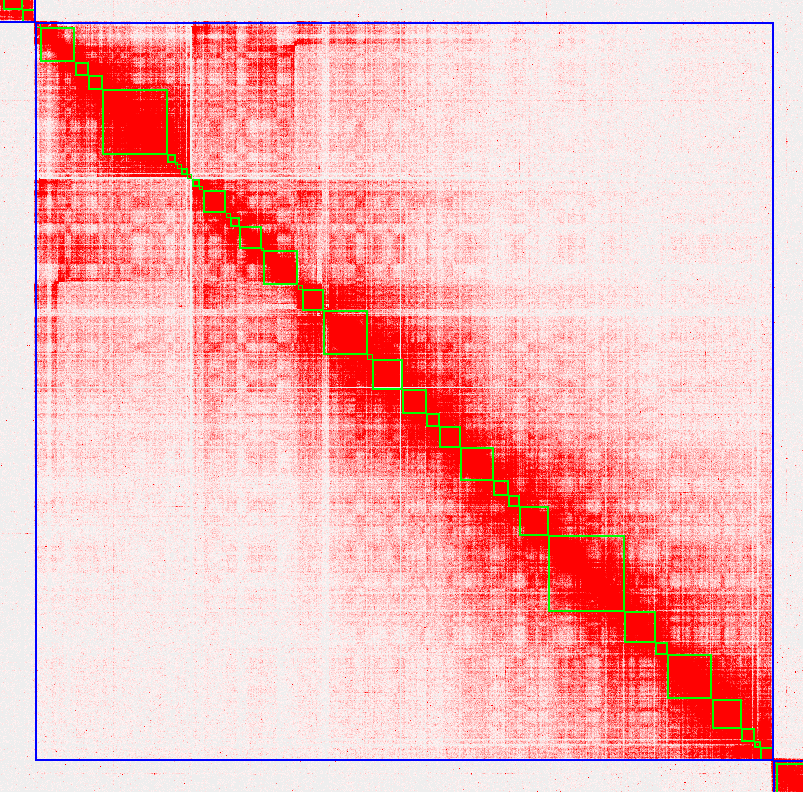
# 4.去除杂合
可以看到有的相邻区块之间没基本有互作,但是与周围区域互作良好,这种情况下这两个区块就是两段杂合区域,原始热图如下所示:
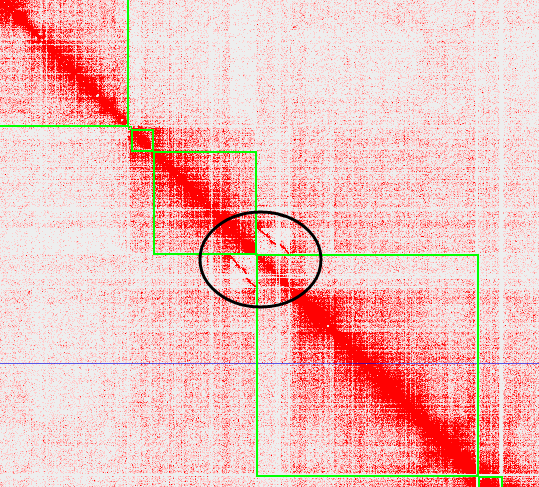
截断取出较短的contig中的杂合区域,挪到热图尾部,建议去杂合最后处理,将杂合区域统一放到一个scaffold方便处理
